Ecosyste.ms: Awesome
An open API service indexing awesome lists of open source software.
https://github.com/xhw205/GPLinker_torch
CMeIE/CBLUE/CHIP/实体关系抽取/SPO抽取
https://github.com/xhw205/GPLinker_torch
Last synced: 2 months ago
JSON representation
CMeIE/CBLUE/CHIP/实体关系抽取/SPO抽取
- Host: GitHub
- URL: https://github.com/xhw205/GPLinker_torch
- Owner: xhw205
- Created: 2022-04-06T09:08:12.000Z (almost 3 years ago)
- Default Branch: main
- Last Pushed: 2022-06-15T03:14:12.000Z (over 2 years ago)
- Last Synced: 2024-08-03T09:07:16.243Z (6 months ago)
- Language: Python
- Homepage:
- Size: 729 KB
- Stars: 205
- Watchers: 4
- Forks: 14
- Open Issues: 6
-
Metadata Files:
- Readme: readme.md
Awesome Lists containing this project
- StarryDivineSky - xhw205/GPLinker_torch
README
## 实体关系抽取_GPLinker
### 介绍
实体关系抽取是文本结构化、构建专业知识图谱的核心 step。
本算法是 [GPLinker](https://kexue.fm/archives/8888) 的 pytorch 复现(简单易懂,杜绝花里胡哨),该方法的核心是:
+ 对输入文本 S={w1,w2,...,wn} 的编码向量以【token-pair】标记方式建模 n×n 大小的词元矩阵,进而做实体识别、实体关系抽取任务。
+ 与之相似的工作有:[TP-Linker](https://arxiv.org/abs/2010.13415)、[multi-head selection](https://arxiv.org/abs/1804.07847)、[Word-pair](https://arxiv.org/pdf/2112.10070.pdf) 等。较之传统的 BIO 序列标注、span 指针网络标注方式,token-pair 建模方式现在是实体关系抽取 sota 必备 schema。
### 数据集
中文医疗信息处理挑战榜 CBLUE 中 CMeIE 数据集,同样是 CHIP2020/2021 的医学实体关系抽取数据集。
### 环境
+ python 3.8.1
+ pytorch==1.8.1
+ transformer==4.9.2
+ configparser
### 预训练模型
[RoBerta-zh-large](https://drive.google.com/file/d/1yK_P8VhWZtdgzaG0gJ3zUGOKWODitKXZ/view)下载
### 运行
请把 config.ini 中对应的【paths】换为你自己的
#### train
```
python main.py
```
#### predict
```
python predict.py
```
### 效果
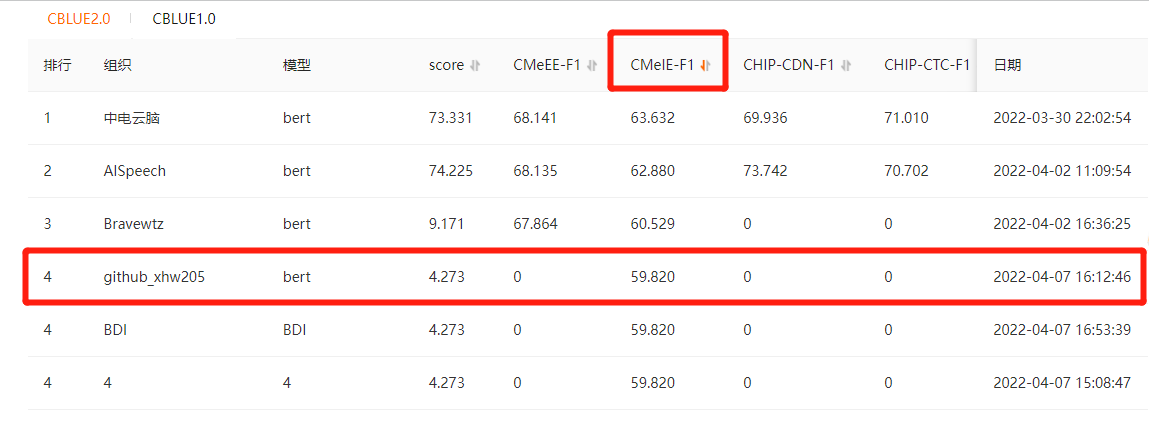
+ 使用医学实体关系抽取数据集,[阿里天池](https://tianchi.aliyun.com/dataset/dataDetail?dataId=95414#4)在线测试F1分数【59.82%】,提交的测试结果在./result文件夹中
【不再提供,可以直接提交的测试结果文件】
+ 之前复现的 CasRel 方法,参考 [DeepIE 仓库](https://github.com/loujie0822/DeepIE) ,在线F1分数为【60.556%】,后续整理开源
> 注意:TOP-1【66.044%】是百度知识图谱团队的 ERNIE ,基本属于吊打其余方法,但是对于在校生、小团队而言,F1分数如果能上【62%】就属于非常非常好了
+ 注意最新的 CBLUE 打榜,需要把生成的 CMeIE_test.json 后缀改为 jsonl,再压缩提交
### TODO
+ 训练过程未根据验证集的F1分数保存最优模型,直接用的最后一个epoch的权重,有需要的自行实现就好了
+ 把globalpointer 替换 Efficient-GlobalPointer,torch的源码本人都已经公布,自行实现就好